清华大学自动化系在合成生物学基因调控序列人工智能设计领域取得进展
2024/03/29
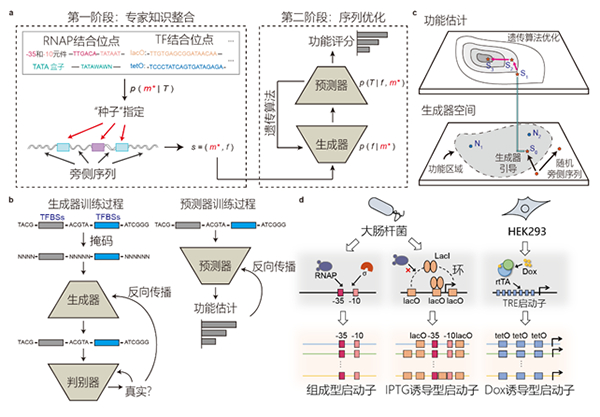 |
图 知识引导与数据驱动相融合的启动子辅助设计方法
在国家自然科学基金项目(批准号:62250007、62225307、61721003)等资助下,清华大学自动化系汪小我教授团队提出了一种将专家知识与大数据学习相融合的合成启动子人工智能辅助设计方法DeepSEED。研究成果以“使用DeepSEED进行侧翼序列深度改造实现高效启动子设计(Deep flanking sequence engineering for efficient promoter design using DeepSEED)”为题,于2023年10月9日发表于《自然•通讯》(Nature Communications)期刊上。论文链接:https://www.nature.com/articles/s41467-023-41899-y。
人类专家擅长从小样本中发现强特征,而深度学习模型可以从大量数据中提取隐含的弱模式。基因启动子是决定基因在什么条件下以何种程度进行转录表达的DNA调控序列。设计具有特定功能的合成启动子是构建人工基因系统的基础,在代谢工程、基因治疗等领域有广泛用途。过去,对启动子序列的设计改造主要集中在修改具有显著模式特征的转录因子结合位点(TFBS)序列上,而忽视了连接不同TFBS之间的侧翼序列。尽管有证据表明侧翼序列对启动子活性具有一定影响,但其特征信号弱且规律复杂,难以被归纳总结为明确的知识和设计准则。
团队针对启动子侧翼序列维度高、模式复杂、在设计中被长期忽视的问题,提出了一种人工智能辅助的侧翼序列优化方法DeepSEED。如图所示,该方法基于知识引导与数据驱动融合的智能设计策略,一方面利用专家知识定义与启动子功能相关的强特征模式作为“种子”,另一方面利用深度学习从大量数据中学习侧翼序列满足的弱特征复杂约束,基于条件式生成模型和进化计算来优化侧翼序列与“种子”的匹配关系,以实现对启动子序列的整体优化提高其转录调控活性。该方法成功应用于细菌和哺乳动物细胞内多类启动子的优化设计,细胞实验验证了其有效性,获得了一批序列多样性强、功能优于天然序列的全新启动子。
该研究提出的知识引导与数据驱动相融合的合成启动子智能设计策略,为解决功能启动子设计中维度高、样本小的核心难题提供了新思路,为合成生物学研究提供可用于旁侧序列优化和多类型启动子设计的人工智能辅助工具。
文章来源国家自然科学基金委员会,分享只为学术交流,如涉及侵权问题请联系我们,我们将及时修改或删除。
-
2026年第八届计算机科学与技术在教 109

-
2026年3月高含金量国际学术会议合 88

-
2026资源、化学化工与应用材料国际 1265

-
2026年人工智能教育技术与数据科学 246

-
2026年图像处理与数字创意设计国际 1041

-
2026年机械工程,新能源与电气技术 5524

-
2026年材料科学、低碳技术与动力工 1312

-
2026年第二届无线与光通信国际会议 2080

-
第七届多组学科研与临床应用大会 02-05

-
2026年制药学、生物技术与植物学国 02-04

-
2026年节能技术、低碳发展与可持续 02-04

-
2026年导航控制、传感技术与卫星应 02-04

-
2026年经济转型与商业文化国际学术 02-04

-
2026年体育科学、运动健康与创新管 02-04

-
2026创意城市、设计与社区建设国际 02-04

-
2025年两院院士增选有效候选人4066

-
2025最新JCR分区及影响因子10070

-
好学术:科研网址导航|学术头条分5098

-
2025年国际期刊预警名单发布!5129

-
2025年中科院期刊分区表重磅发16711

-
中国科协《重要学术会议目录(2010250

-
吉林大学校长张希:学术会议中的提6245

-
bbrc期刊,bbrc期刊处于S02-03

-
siam期刊,siam期刊发的最02-03

-
了望期刊,瞭望期刊02-03

-
共享经济期刊,共享经济学术论文02-03

-
期刊代理网,从哪里找期刊代理02-03

-
light期刊,Light期刊综02-03

-
传承 期刊,传承期刊投稿点评02-03

-
期刊勘误,期刊勘误时会索要原始数02-03

-
北京永康中医药研究院 2280

-
湖北研学文化发展有限公司 8189

-
GRESGRE 8341

-
重庆燕泰会议服务有限公司 18261

-
亚太科学与工程研究所 2261

-
新村街二七二社区 18143

-
武汉工程大学 21396

-
湖北新文盛会务有限公司 23259

-
博锐国际展览有限公司 2237

-
上海电子信息 23348

-
WILL 2239

-
北京交通大学 2438

-
清华大学持久性有机污染物研究中心 2352

-
江苏省南京市 2608

-
中国人力资源开发杂志 23173

-
北京联合大学信息学院 23257

-
哈尔滨医科大学 2600

-
华中科技大学 21354

-
Global Science & 24440

-
中国石油天然气股份有限公司石油化 8222



















 688
688














































