中国农业大学动科学院方美英教授团队在《自然·遗传》发表猪T2T参考基因组 深入解析猪群体结构与体型选择的遗传基础
2025/12/17
中国农业大学动物科学技术学院方美英教授团队在国际知名学术期刊《自然·遗传》(Nature Genetics)发表开创性研究论文《公猪T2T基因组组装为群体结构与体型选择研究提供了新见解》(Telomere-to-telomere genome assembly of a male pig provides insight into population structure and selection for body stature)。该研究首次完成了中国五指山小型猪端粒到端粒无间隙基因组组装(T2T-pig1.0),其中包含完整的Y染色体和着丝粒区域。
家猪(Sus scrofa)自约一万年前被驯化以来,已成为全球重要的农用动物和生物医学研究模式动物。然而受测序技术限制,目前猪参考基因组在重复序列富集区域仍存在数百个缺口尚未解析,如着丝粒和Y染色体异染色质区。此外,由于缺乏高质量、完整的家猪基因组资源,其基因组进化及决定独特性状的关键突变仍缺乏深入理解。
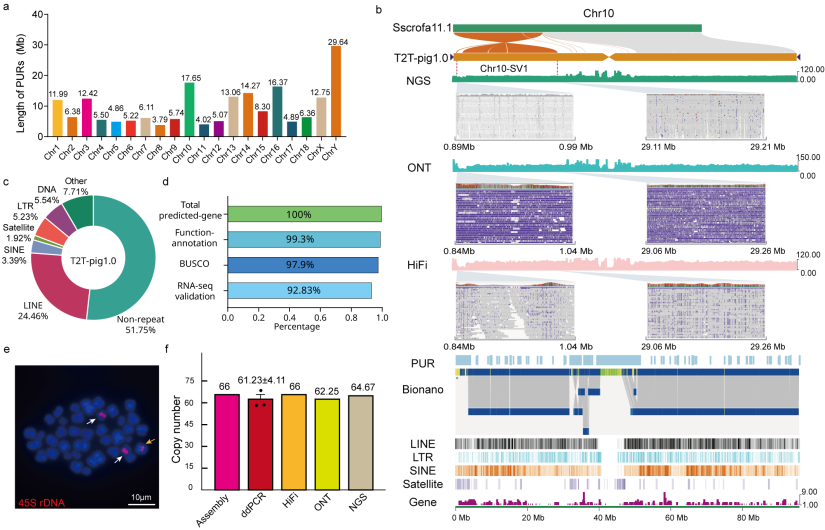
本研究整合169.8 Gb超长ONT(65.31×)、192.4 GbPacBio HiFi(73.98×)、1113.8 GbBionano光学图谱数据、267 GbHi-C数据(100×),以及133.7 Gb二代测序(50×)数据,构建了一个2.63 Gb,QV值达到51.41的无缺口T2T基因组(T2T-pig1.0)。与现有参考基因组Sscrofa11.1相比,T2T-pig1.0新增解析区域194.42 Mb,其中包含1189个基因。
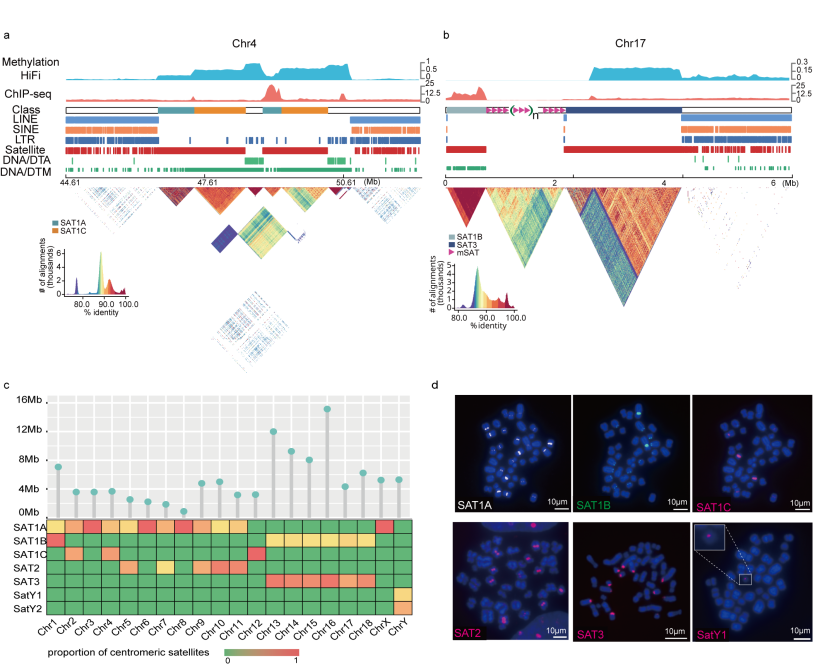
综合利用染色质免疫共沉淀测序(ChIP-seq)、HiFi甲基化信号以及着丝粒卫星序列,本研究鉴定出总长度约为107.3 Mb(占T2T-pig1.0基因组的4.0%)着丝粒区域。重复序列注释显示,五种着丝粒卫星重复单元(SAT1–SAT3、SatY1和SatY2)在着丝粒区域中占主导地位;这些卫星单元表现出明显的染色体分布偏差。其中,SAT3仅定位于六条端着丝粒染色体的着丝粒区域,并与微卫星序列“AGAAT”(mSAT)和SAT1B呈现独特的端着丝粒特异分布模式:“telomere-SAT1B-(mSAT)n-SAT3-q_arm”。此外,研究发现了猪着丝粒中存在独特的DNA转座子和LTR入侵。上述结果为猪着丝粒图谱绘制和结构特征解析提供了新的视角。
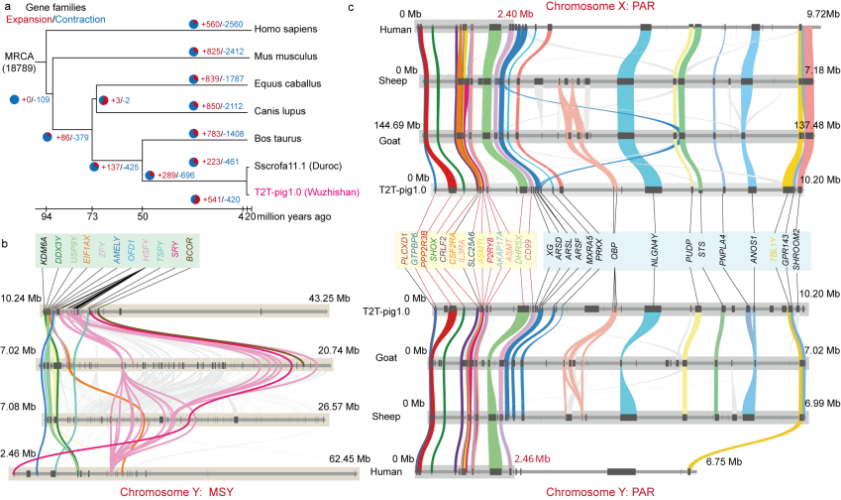
T2T-pig1.0实现完整无缺口Y染色体组装,长度为43.25 Mb,相比Sscrofa11.1版本的ChrY新增了29.64 Mb此前未解析序列;同时鉴定出两种新的ChrY特异着丝粒重复SatY1(5660 bp)和SatY2(2472 bp)。跨物种比较分析揭示,五指山猪与杜洛克猪约在210万年前分化。T2T-pig1.0中有541个基因家族扩增,这些扩增基因显著富集于“细胞大小负调控”和“器官生长负调控”等通路,可能与五指山猪小体型性状相关。此外,猪、人、山羊和绵羊四种大型动物的T2T性染色体比较表明,不同物种的ChrX与ChrY在PAR和MSY区域维持较高共线性,可能具有共同进化起源。
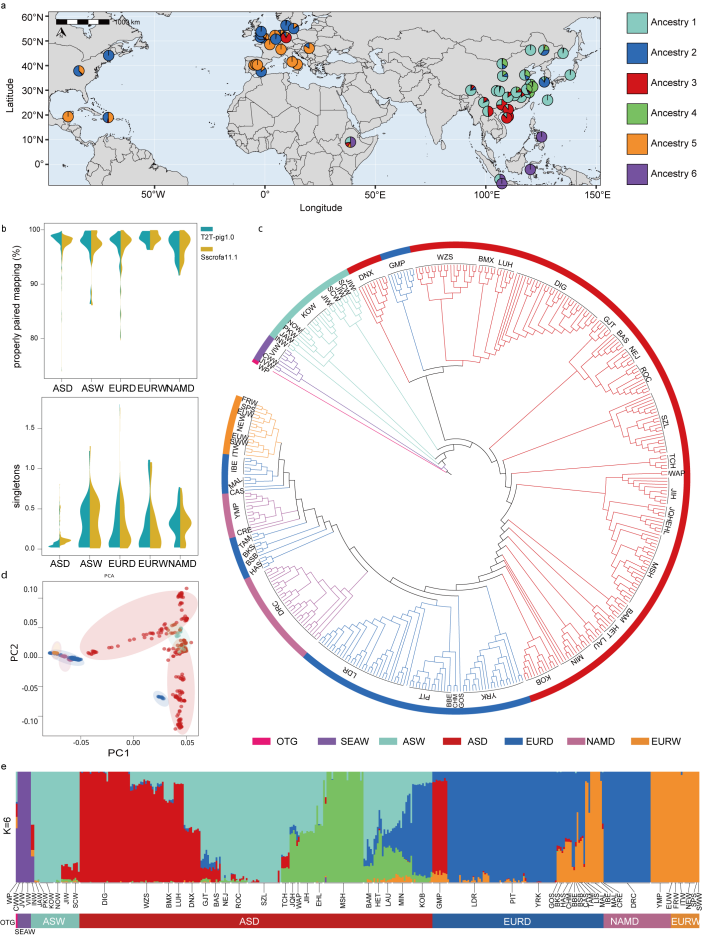
基于T2T-pig1.0参考基因组,本研究对来自60个品种、覆盖亚洲、欧洲和北美的407份全基因组测序数据进行了群体遗传分析。与Sscrofa11.1相比,T2T-pig1.0的比对准确性显著提升,错配率和未配对读段数量均减少。以T2T-pig1.0为参考共检测到约1785万个SNPs,其中1.9%位于PUR区域。PUR区SNPs在亚洲和东南亚群体中以低频等位基因为主,在欧洲和北美群体中趋于固定。基于这些SNPs构建的系统发育树、PCA及群体结构分析结果揭示,亚洲家猪由三种主要祖源成分构成,且东北亚猪群中存在欧洲家猪的遗传成分;基于T2T-pig1.0的分析更精准地揭示了家猪群体的遗传结构及亚洲猪群间的基因渗入关系。
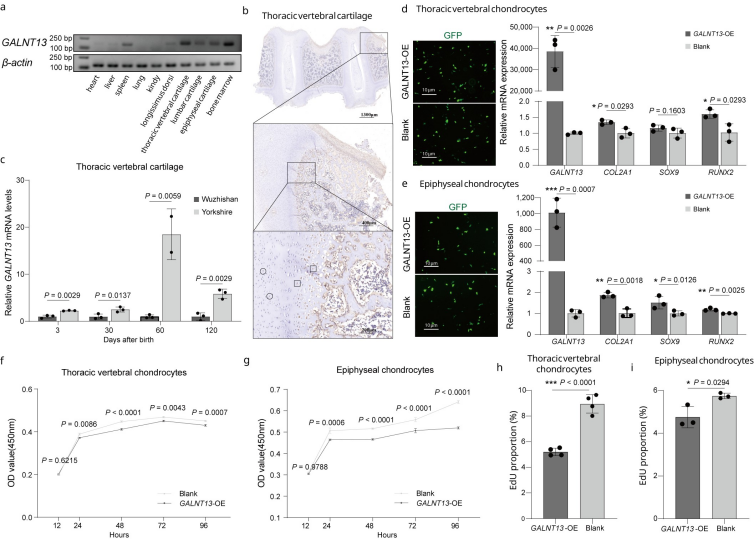
基于T2T-pig1.0基因组鉴定的SNPs进行选择信号分析,通过FST共鉴定到2956个选择区域、657个候选基因(其中280个位于PUR区域)。PUR基因GALNT13在所有比较中均表现出显著选择信号。进一步功能研究发现,GALNT13在猪骨相关组织(胸椎软骨、腰椎软骨、骺软骨及骨髓)中高表达,且在小型猪中表达量显著降低;该基因过表达可促进软骨细胞分化(上调COL2A1、SOX9和RUNX2表达)、抑制其增殖,表明GALNT13通过调控软骨细胞的增殖与分化进程影响体长发育,进而参与猪体型形成。
本研究为猪基因组研究提供了关键基础数据,研究成果对猪品种改良具有重要指导价值,同时拓展了猪作为生物医学模式动物的应用前景。
中国农业大学为论文唯一通讯单位,中国农大动科学院博士后雒亚彪,博士生黄宁和查程万为共同第一作者,方美英教授和贾善刚老师为共同通讯作者。吴常信院士为本研究提供了实验室平台支撑,李宁教授、姜志华教授在文章修改过程中给予了建设性指导。海南省农业科学院畜牧兽医研究所提供测序样本。本研究获国家自然科学基金区域联合基金重点项目(U22A20508)、国家重点研发计划、拼多多-中国农业大学研究基金等项目资助。
文章来源中国农业大学,分享只为学术交流,如涉及侵权问题请联系我们,我们将及时修改或删除。
-
2026年第八届计算机科学与技术在教 109

-
2026年3月高含金量国际学术会议合 88

-
2026资源、化学化工与应用材料国际 1265

-
2026年人工智能教育技术与数据科学 246

-
2026年图像处理与数字创意设计国际 1041

-
2026年机械工程,新能源与电气技术 5524

-
2026年材料科学、低碳技术与动力工 1312

-
2026年第二届无线与光通信国际会议 2080

-
第七届多组学科研与临床应用大会 02-05

-
2026年制药学、生物技术与植物学国 02-04

-
2026年节能技术、低碳发展与可持续 02-04

-
2026年导航控制、传感技术与卫星应 02-04

-
2026年经济转型与商业文化国际学术 02-04

-
2026年体育科学、运动健康与创新管 02-04

-
2026创意城市、设计与社区建设国际 02-04

-
2025年两院院士增选有效候选人4066

-
2025最新JCR分区及影响因子10070

-
好学术:科研网址导航|学术头条分5098

-
2025年国际期刊预警名单发布!5129

-
2025年中科院期刊分区表重磅发16711

-
中国科协《重要学术会议目录(2010250

-
吉林大学校长张希:学术会议中的提6245

-
bbrc期刊,bbrc期刊处于S02-03

-
siam期刊,siam期刊发的最02-03

-
了望期刊,瞭望期刊02-03

-
共享经济期刊,共享经济学术论文02-03

-
期刊代理网,从哪里找期刊代理02-03

-
light期刊,Light期刊综02-03

-
传承 期刊,传承期刊投稿点评02-03

-
期刊勘误,期刊勘误时会索要原始数02-03

-
中国化工学会培训中心 2257

-
深圳市安视佳电子监控摄像机厂家 18276

-
南方医科大学 2361

-
上海创蓝文化传播有限公司 8169

-
亚泰RFID技术协会 23431

-
电子科技大学计算机学院国家级计算 2448

-
盐城工学院东校区 21384

-
西北大学 24306

-
中国传媒大学 23190

-
花蓮東華大學 2227

-
北京沃特咨询有限公司 8839

-
华大基因研究院 18218

-
北京科技大学 23231

-
重庆邮电大学 23489

-
梵净山红豆杉开发有限公司 18208

-
上海巴典尼球阀厂 18211

-
大秦国际--新疆西部游旅行社会议 18506

-
广州悦雅会议服务有限公司 18402

-
同研中心 18257

-
中华医学会 21564



















 95
95