高歌课题组提出基因丢失鉴定新方法
2024/04/07
作为细胞中基本的功能单位,蛋白编码基因(Protein-coding genes,以下简称基因)可以通过转录-翻译过程指导合成对于生命活动至关重要的蛋白质,进而影响生命体的生理/病理性状。因此,基因的演化与生物体的表型演化息息相关,是介导物种特异性表型及其环境适应的重要动力之一。在演化过程中生物体可以获得新的基因来执行新的功能;因此,自上世纪后期至今,新基因(Gene Birth)已成为相关领域的研究热点。另一方面,万物皆有生有灭,与不断涌现的新基因相对,现有的蛋白编码基因也可能因突变等失去原有的功能并进而从基因组中丢失。然而,受限于可用数据等多方面因素,对基因丢失(Gene Loss)及其影响长期以来尚缺乏系统研究。
近日,北京大学生物医学前沿创新中心(BIOPIC)、北京未来基因诊断高精尖创新中心(ICG)、北京大学生命科学学院生物信息中心(CBI)、蛋白质与植物基因研究国家重点实验室高歌课题组在期刊Molecular Biology and Evolution上发表了题为“Genome-wide identification of gene loss events suggests loss relics as a potential source of functional lncRNAs in humans”的生物信息学论文,在前期工作基础上提出了新的基因丢失鉴定方法LOST&FOUND,并基于该方法对人类中的基因丢失及其影响进行了系统解析。
为精准鉴定基因丢失事件,高歌团队提出了新的基因丢失鉴定方法LOST&FOUND。LOST&FOUND的具体处理流程如图1所示。LOST&FOUND结合了全基因组比对以及多物种中的直系同源基因数据,并运用最大简约法等处理方法来对基因丢失进行鉴定识别。基于多物种直系同源基因关系的推断可使LOST&FOUND避免将参考物种中的基因获得事件错误识别为待研究物种中的基因丢失事件。而全基因组比对的使用则可充分考虑基因及其上下游区段的同源性,有利于LOST&FOUND识别由大片段删除等方式造成的基因丢失事件。
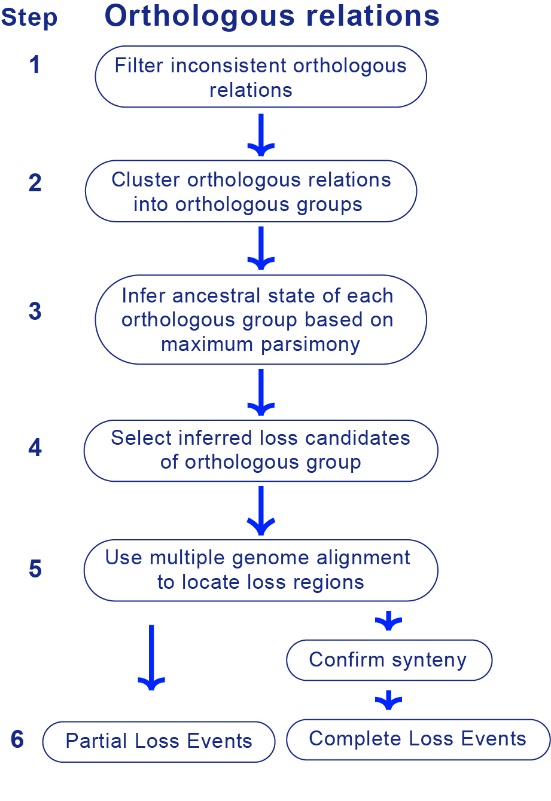
图1
基于该方法,团队在人类基因组中共鉴定到155个基因丢失事件。其中,有88个基因丢失事件在人类基因组内含有同源区段残骸(relics)。有趣的是,通过将基因丢失残骸与长非编码RNA区段进行比较,团队发现,在人类基因组中,有33个基因丢失事件的发生与长非编码RNA的起源相关,并将该部分通过基因丢失起源的长非编码RNA命名为derived lncRNA(图2 A-B)。

图2
综合多方面的注释,团队发现,derived lncRNA与其它长非编码RNA不同,其往往有着更高的表达量、更低的组织表达特异性、更长的转录本结构以及更强的序列保守性(图3 A-F)。基于GWAS、共表达、已有实验检索等功能性分析的结果则表明,这部分derived lncRNA与生长发育、免疫、生殖以及抑癌作用等过程的调控均有关系。同时,超过一半的derived lncRNA均受到了正选择的作用。
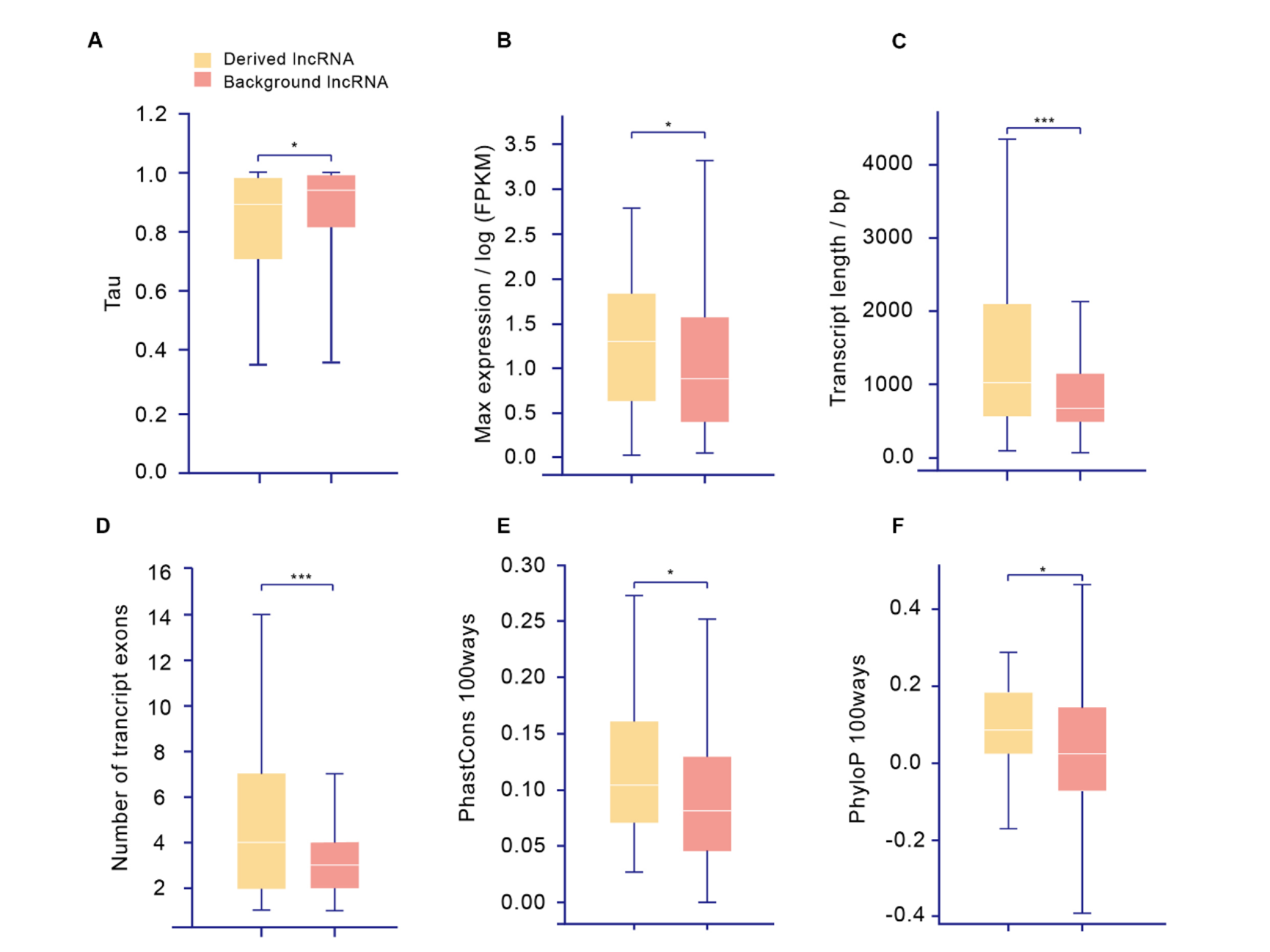
图3
高歌团队开发了新的基因丢失鉴定方法并通过该方法系统性研究了人类基因组中基因丢失的发生与影响。值得注意的是,高歌团队在人类基因组中发现了33个基因丢失事件与长非编码RNA的起源相关,且这部分长非编码RNA可能具有重要功能。结合前期已发表工作1-3,这些结果提示在多个物种中,演化过程中古老的蛋白编码基因有可能“再生”为新的长非编码RNA来发挥作用,在基因生-死、编码-非编码等看似对立的概念之间建立起了有趣的联系。
高歌的博士生温正扬为该论文第一作者,高歌为该论文通讯作者。该研究得到了蛋白质与植物基因研究国家重点实验室、北京未来基因诊断高精尖创新中心等的资助。计算分析工作于北京大学高性能计算校级公共平台和北京大学太平洋高性能计算平台完成。
参考文献:
1 Duret, L., Chureau, C., Samain, S., Weissenbach, J. & Avner, P. The Xist RNA gene evolved in eutherians by pseudogenization of a protein-coding gene. Science312, 1653-1655 (2006).
2 Zhao, Y. et al. Identification and analysis of unitary loss of long-established protein-coding genes in Poaceae shows evidences for biased gene loss and putatively functional transcription of relics. BMC Evol Biol15, 66 (2015).
3 Hezroni, H. et al. A subset of conserved mammalian long non-coding RNAs are fossils of ancestral protein-coding genes. Genome Biol.18, 162 (2017).
文章来源北京大学新闻网,分享只为学术交流,如涉及侵权问题请联系我们,我们将及时修改或删除。
-
2025年智能光子学与应用技术国际学 06-10

-
2025年8月优质学术会议推荐 589

-
2025年第十二届能源与环境研究国际 400

-
2025年机械工程,新能源与电气技术 559

-
2025年计算机科学、图像分析与信号 553

-
2025年材料化学与燃料电池技术国际 493

-
第七届精神病学国际大会(CP 202 08-01

-
第七届老龄化与老年医学国际学术会议( 08-01

-
第十四届心理学与健康国际学术研讨会( 08-01

-
第七届测绘与地理信息国际研讨会(CS 08-01

-
第七届大气与海洋科学国际研讨会(SA 08-01

-
第十一届地质灾害研究与防治国际学术会 08-01

-
第十六届地质和地球物理学国际会议(I 08-01

-
第七届细胞科学与再生医学国际研讨会( 08-01

-
第七届合成生物学与生物医学国际研讨会 08-01

-
2025最新JCR分区及影响因子1939

-
好学术:科研网址导航|学术头条分468

-
《时代技术》投稿全攻略:一位审稿499

-
2025年国际期刊预警名单发布!600

-
2025年中科院期刊分区表重磅发3957

-
中科院已正式发布2024年预警期861

-
2025年度国家自然科学基金项目727

-
中国科协《重要学术会议目录(202733

-
2024年国家自然科学基金项目评1138

-
2024年JCR影响因子正式发布1214

-
吉林大学校长张希:学术会议中的提1391

-
SCI论文插图全攻略:从规范解析08-01

-
国际学术会议参加经验是怎么样的呢08-01

-
掠夺性会议是怎么进行判断的呢?—08-01

-
SCI论文投稿费怎么交?202408-01

-
长白山明华会议 21093

-
International As 8160

-
意沃企业管理咨询 8056

-
郑州大学第一附属医院眼科 20940

-
赛诺瑞 7917

-
中 华 医 学 17971

-
上海浩韵文化传播有限公司 23088

-
武汉泰迪智慧科技有限公司 22913

-
个体 1948

-
北京建筑工程学院 20878

-
亚太科学与工程研究所 24044

-
上海商图信息咨询有限公司 7931

-
上海荣格展览有限公司 21019

-
北京邮电大学 2018

-
长安大学 2054

-
全国医药技术市场协会 23920

-
湖北学而升文化传播有限公司 24065

-
广州市金晔展览有限公司 7998

-
国际工学技术出版协会 7877

-
中国地震局工程力学研究所 18063



















 360
360













































